ABI-SOLID原理: 非PCR连接合成法测序
ABI-SOLID的基本原理是非PCR连接合成法测序(与边合成边测序思想不同):它不以待测序列为模板进行PCR扩增测序,而是利用DNA连接酶将一段寡核苷酸探针连接至模板上进行测序。
在该测序方法中,需明白模板序列(待测序列)、靶序列(8-9nt寡核苷酸探针单链序列,其上某位置的核苷酸已知,且其5'端进行对应的荧光素标记)和锚定序列(序列已知的一段DNA,与未知模板序列配对结合,起到模板固定的作用)几个概念。
首先,锚定序列以互补配对方式结合至待测序列模板上。随即,靶序列与模板序列进行匹配。当靶序列与模板序列匹配时,DNA连接酶可识别特定位置的核苷酸,从而将靶序列与锚定序列连接在一起。反之,当靶序列不匹配时,DNA连接酶很难进行靶序列与锚定序列的连接。

在实际测序工作中,靶序列是多组一系列寡核苷酸探针。这些寡核苷酸探针具有这样的特点:某一固定位置的碱基已知,其5'端进行相应的荧光标记。
简单地说,若该探针3'端第一个碱基已知(其他位置的碱基序列为随机)为A,那么此时在其5'端进行确定的荧光标记(蓝色)。若第一个已知碱基为C,5'端标记为黄色。以此类推,如下图:

随后,将这组靶向探针与模板进行配对。若靶向探针与模板配对,此时DNA连接酶即可将该探针序列与锚定序列进行连接。如此,可知锚定序列5'端其待测序列的第一个碱基是什么碱基了。
其后,升温变性,锚定序列将锚定至新的未知序列上,靶向探针与模板配对,连接反应再次发生,从而获知未知序列相应位置的核苷酸。
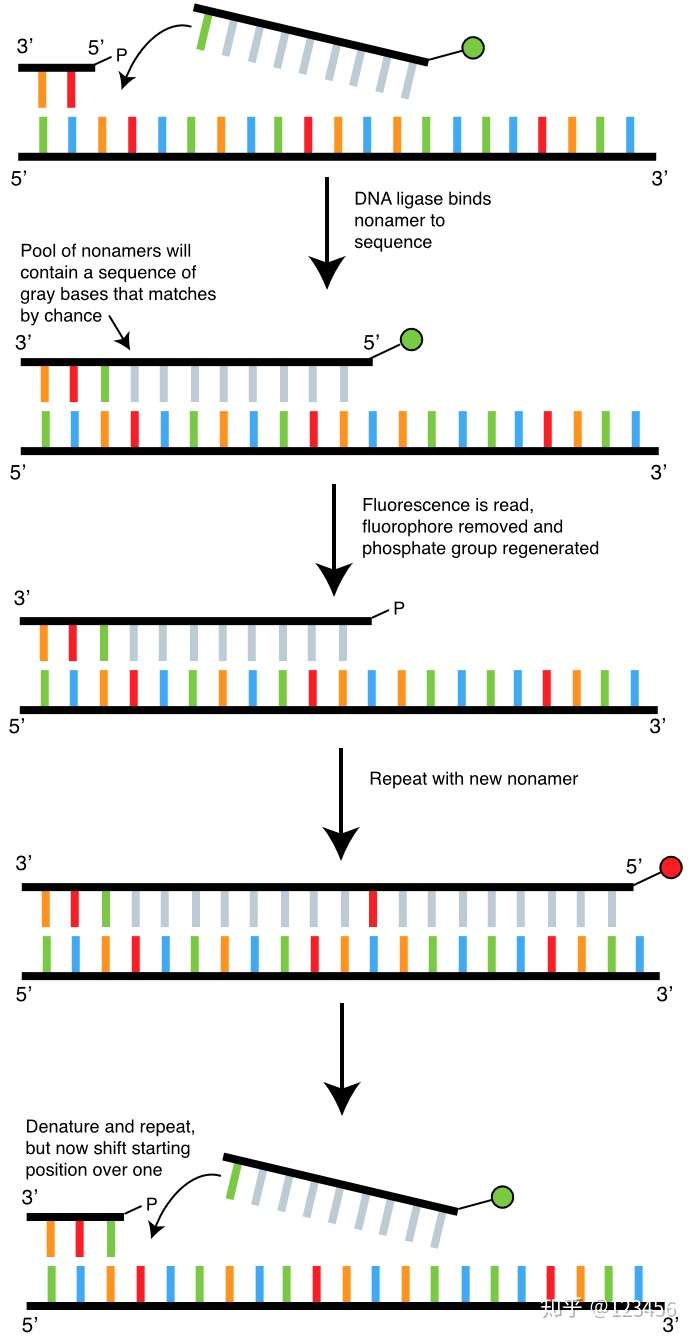
随着每一组靶向探针与锚定序列连接序列产生,未知序列的序列信息逐一得到破解。需要注意的是:
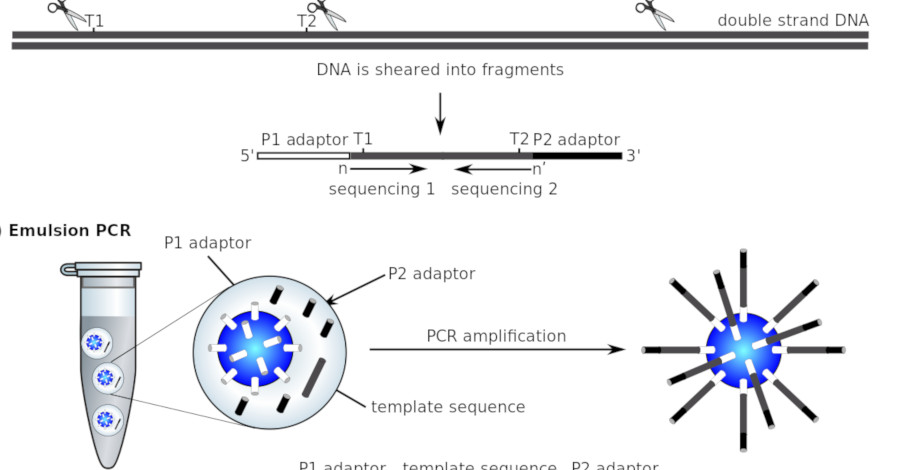
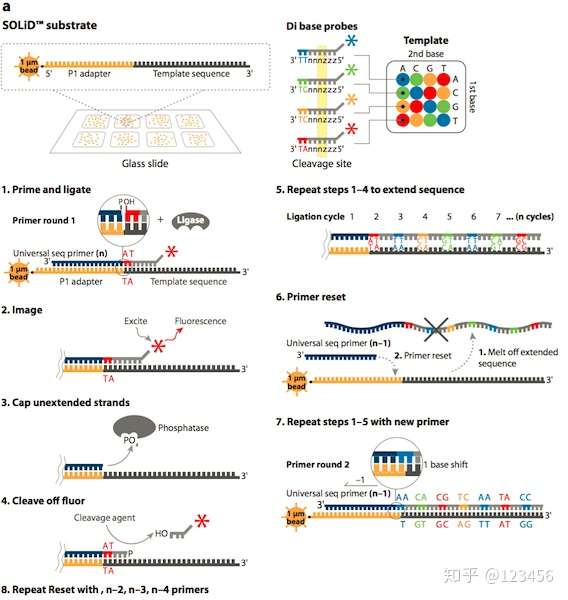
1. 上述待测模板序列实际上是待测长DNA模板中的一小段,其产生仍然需要采用油包水PCR方式进行扩增(测序文库)。扩增得到的待测片段通过杂交方式,与共价连接到磁珠上的Adapter其P1引物配对固定。因此,SOLID测序反应实际上是在在共价结合待测片段模板的玻璃磁珠表面进行的一系列连接反应(每个磁珠都代表一段待测序列)。每个磁珠是Solid测序的最小反应单元。
2. “锚”定序列与待测模板P1引物互补:P1引物一般有五种,长度相同。但在P1引物区域位置相差一个碱基(n,n-1,n-2,n-3,n-4),均含有5'端磷酸,用以保证连接反应的进行。
3. 寡核苷酸探针其序列总长度为8-9nt,其中3'端有1-2个碱基序列是已知的,其他位置为通用碱基(标记为“z”),可与待测序列上的任意位置序列互补配对。在探针的5'端,一般用四种荧光进行标记,分别标记A、G、C、T。
4. 测序过程重复5-7轮后可测得待测序列全长,每次重复可通过探针切割,实现测序起始位点的改变,从而实现不同起始位置的测序连接反应。
SOLID非PCR连接合成法测序具有一下特点:
1. 因SOLID探针已知序列长度为2(且只有四种荧光标记),因此测序结果会产生简并性(即AT、GC、CG和TA荧光标记相同),故某个探针错配(荧光颜色错误)会导致后续连锁反应,导致多个错配连续发生,因此,该方法需采用参考序列进行校正。
2. 该方法文库制备相当繁琐,价格昂贵且,成本极高。
3. 与边合成边测序方法比较,,本方法具有更高准确度。特别是,当覆盖度进行到15×,其准确度可达到99.99%。
原文来自:https://zhuanlan.zhihu.com/p/52515440(已做修改)
其他资料:Revolutionary Technology SOLID Platform(PDF)